Selenophosphate Synthetase 1 (SPS1)
SPS1 es una proteïna de maquinària que no es troba anotada a selenoDB per a Myotis lucifugus. És per això, que hem hagut d'utilitzar la query de la proteïna d'Homo sapiens.
El blast ens dòna set hits, el primer d'ells en el mateix sentit a l'anotació (+) i els altres sis en sentit contrari a l'anotació (-). D'entre els set hits, l'scaffold escollit és KB116581.1, ja que, tot i que la seva homologia no és tant alta respecte als altres hits, la seva puntuació de tcoffee és molt més elevada.
L'exonerate ha predit una sola proteïna amb 7 exons i 6 introns, i una raw score de 1617. Pel que fa al genewise, ha predit també una sola proteïna amb 7 exons i 6 introns i un score de 680.46 bits.
Tenint com a referència l'altra puntuació del tcoffee podem hipotetitzar que la estructura està ben conservada.
Al ser una proteïna de maquinària i no contenir selenocisteïnes en la seva seqüència, no trobem estructures SECIS ni Seblastian mitjançant la predicció per Secisearch 3, cosa que ens valida que SPS1 no es tracta d'una selenoproteïna en Myotis davidii.
Que no s'hagi trobat en Myotis lucifugus i si en Myotis davidii pot indicar que aquesta s'ha perdut en aquesta espècie. Tot i amb això, donat que és una proteïna de maquinària essencial, conservada en la resta de vertebrats, podem hipotetitzar que es tracti d'un problema en l'anotació.
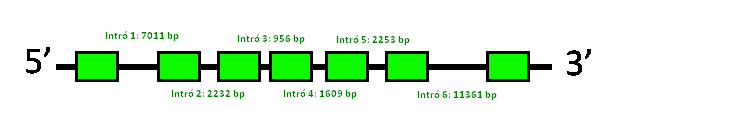
Selenophosphate Synthetase 2 (SPS2)
SPS2 és una selenoproteïna que no es troba anotada a selenoDB per a Myotis lucifugus. és per això que hem hagut d'utilitzar la query de la proteïna de Homo sapiens.
El blast ens dóna set hits, tots ells en sentit contrari a l'anotació (-) menys el 3r hit amb major homologia que té sentit +. D'entre els set hits, l'scaffold escollit és KB189536.1; ja que presenta la homologia més alta de tots els hits i una puntuació de tcoffee molt elevada.
L'exonerate ha predit una sola proteïna amb 1 exó i sense introns, i una raw score de 1617. Pel que fa al genewise, ha predit també una sola proteïna amb 1 exó i sense introns, i un score de 740.39 bits.
L'estructura predita amb l'exonerate presenta un codó stop a la mateixa posició que la selenoproteïna de la query (aminoàcid 60). L'estructura predita pel genewise també ens senyala amb una X, la selenocisteïna en el mateix lloc que la query humana. Deduïm així, que la selenoproteïna està ben conservada. Pel que fa al tcoffee, com ja hem dit, trobem una puntuació alta que reafirma la nostra hipòtesi de que la estructura està conservada.
Tot i que la nostra proteïna conté una selenocisteïna, no trobem estructures SECIS mitjançant la predicció per Secisearch 3, i el Seblastian no ens la prediu. Hipotetitzem que es tracta d'un error en la predicció de la selenoproteïna, ja que SPS2 ès una proteïna essencial de maquinària (ès el donador de la selenocisteïna). Igualment, el fet de no trobar-lo en Myotis lucifugus ha de ser un problema d'anotació, ja que la trobem en tots els vertebrats.